![001]()
Esta es la foto 51, una foto tomada por Rosalind Franklin y su ayudante Raymond Gosling con la que se pudo discernir el secreto de la estructura del ADN. El modelo fue propuesto por Watson y Crick gracias a que tuvieron acceso a esta foto en una jugada un tanto criticable.
Esta entrada tiene por objeto describir cómo se puede deducir la estructura del ADN, una doble hélice, a partir de esa foto. La historia asociada no deja de ser interesantísima y es una obligación dedicar unos minutos a conocerla y a homenajear a la mujer que la hizo posible, Rosalind Franklin. Os dejo un vídeo donde se hace un breve esbozo de su vida y obra:
Y aquí el vídeo de lo que vamos a desarrollar en esta entrada, la deducción de la estructura del ADN a partir de la foto 51:
Si al acabar esta entrada crees que molaría tener un detalle con este vuestro humilde autor me conformaría con tener vuestro voto en los premios bitácoras para el podcast @Los3_Chanchitos. Este es un podcast de ciencia y cultura en general (3chanchitos.es), con mejor o peor humor, del que formo parte. Si deseas votar en señal de infinita gratitud solo tienes que pulsar aquí abajo:
![Votar en los Premios Bitacoras.com]()
Basta de peticiones, vayamos al lío.
Ondas electromagnéticas y espectro
Generalmente para “ver” algo utilizamos luz. Pero como todos hemos escuchado alguna vez la luz es una onda electromagnética. Estas ondas electromagnéticas son unos campos eléctricos y magnéticos que van propagándose ondulatoriamente según dictan las consecuencias de las ecuaciones del electromagnetismo de Maxwell.
![002]()
Como buena onda tiene varias características de interés. Para lo que nos ocupa nos interesa especialmente una, la longitud de onda, ![\lambda \lambda]() . Esta es la distancia entre dos picos de la onda, dos valles o cualquier par de puntos en los que la onda se encuentre en el mismo estado de oscilación.
. Esta es la distancia entre dos picos de la onda, dos valles o cualquier par de puntos en los que la onda se encuentre en el mismo estado de oscilación.
La longitud de onda nos permite clasificar las ondas electromagnéticas en función de su longitud de onda. La clasificación se denomina espectro electromagnético.
![003]()
Las ondas electromagnéticas de menor a mayor longitud de onda se clasifican en:
Rayos Gamma
Rayos X
Ultravioleta
Visible
Infrarrojo
Microondas
Ondas de Radio
Los rayos gamma tienen longitudes de onda de 0.000000000001 m y las ondas de radio pueden tener longitudes de onda de kilómetros. Evidentemente, las divisiones entre estas clasificaciones no son estrictas sino que hay regiones en las que las distinciones no se aprecian entre una clase y otra. Es simplemente una clasificación que nos ayuda a etiquetar las ondas electromagnéticas en función de su longitud de onda.
Rayos X
En lo que sigue nos vamos a concentrar en los rayos X. Estos rayos X son ondas electromagnéticas que tienen longitudes de onda comparables a los tamaños de los átomos. Su longitud de onda es muy pequeña y como veremos en lo que sigue es lo que nos permite “mirar” dentro de los materiales para “ver” su estructura atómica o molecular.
El rango de la longitud de onda de los rayos X oscila entre 0.1 y 10nm. El nanómetro, nm, equivale a 0.000000001m.
La luz que llamamos visible está en el rango de los 400 a los 800nm aproximadamente. Es ciertamente una ventana muy pequeña de todo el posible espectro electromagnético.
Generación de las ondas electromagnéticas
Según la teoría electromagnética de Maxwell una onda electromagnética se genera cuando sometemos a una carga eléctrica a una aceleración. Así que si tenemos una carga eléctrica describiendo un movimiento oscilatorio estará acelerando y emitiendo ondas electromagnéticas.
![004]()
Para generar rayos X lo que se suele hacer es lanzar electrones con mucha velocidad sobre un metal. Al entrar al metal estos electrones sentirán la presencia de núcleos y sufrirán un frenado, una aceleración al fin y al cabo, y si ajustamos bien la velocidad o energía de los electrones lanzados tendremos una emisión de rayos X. Existe otro proceso que involucra que estos electrones arranquen electrones de orbitales internos de los átomos y entonces los electrones de capas superiores caigan a ese nivel interno liberando rayos X, dependiendo del material empleado, con una longitud de onda fijada.
Lo que te tiene que quedar claro es que somos capaces de crear haces de rayos X con longitudes de onda conocida.
¿Qué es ver? La resolución
Cuando decimos que vemos un objeto es porque está iluminado y refleja la luz en su superficie y esta alcanza nuestro detector de ondas electromagnéticas, el ojo. Pero nos es bien conocido que no podemos ver cualquier cosa, hay un límite para el tamaño de las cosas que podemos ver con nuestros detectores. Eso es debido a que para poder “ver” hemos de “iluminar” el objeto con una onda electromagnética de una longitud de onda que sea más pequeña que el objeto ya que de otra manera no podremos resolver la forma del objeto. En nuestro entorno no tenemos problemas porque la luz visible está en el rango de los muchos cientos de nanómetros a cerca de los miles de los mismos y los objetos que nos rodean son bastante mayores de esa escala.
Pero observemos esta imagen:
![resolution-wavelength-1]()
En la primera parte de la imagen no podemos resolver la estructura que estamos iluminando porque la longitud de onda es mayor que la separación entre los dos objetos. En la parte de abajo no tenemos problema para ver los dos objetos nítidamente.
Si lo que queremos es observar la estructura atómica o molecular de las cosas que nos rodean hemos de emplear ondas electromagnéticas que tengan una longitud de onda del mismo orden de las distancias atómico-moleculares. Es por eso que hemos de emplear rayos X.
Los rayos gamma son de menor longitud de onda pero esos no nos sirven porque son tan energéticos que interactuarían con los electrones de los átomos y serían capaces de romper enlaces químicos con lo que no nos sirven para la tarea que nos estamos proponiendo realizar.
Dispersión de ondas electromagnéticas
Hay un efecto interesante con el efecto que tienen los rayos X cuando interactúan con electrones. Suponte que tienes un electrón en reposo en una región y haces que pase una onda electromagnética en el rango de los rayos X. Las ondas electromagnéticas son campos eléctricos y magnéticos que oscilan y se propagan por el espacio con lo que una carga eléctrica sentirá sus efectos. En este caso el electrón empezará a oscilar conforme pase la onda:
![005]()
Muy bonito, muy natural. Pero… Ojo, ese electrón es una carga eléctrica y una carga eléctrica emite ondas electromagnéticas cuando está acelerada. Ese electrón está claramente acelerado así que tendrá que emitir ondas electromagnéticas en dicho proceso.
En efecto, ese electrón emite ondas electromagnéticas en el rango de los rayos X en todas las direcciones (casi todas, pero es un detalle que no nos preocupa ahora mismo).
![006]()
El efecto neto es que hemos introducido un haz con una determinada dirección y acabamos con ondas electromagnéticas en todas las direcciones. Tenemos entre manos una dispersión de la luz.
Este fenómeno ocurre más o menos igual cuando los electrones forman parte de un átomo y pasa por ahí un rayo X.
Interferencias
Una de las propiedades más características de las ondas en general es su capacidad para interferir. La interferencia es la superposición de varias ondas (que tengan propiedades parecidas como la longitud de onda) que llegan a un determinado punto del espacio a la vez.
En las interferencias podemos tener dos casos extremos:
1.- Interferencia constructiva — Esto se da cuando las ondas que se superponen llegan en fase, es decir, los picos y valles de las ondas coinciden en la región de superposición. El efecto es que las ondas se refuerzan.
2.- Interferencia destructiva — En este caso las ondas que van a interferir en una región llegan opuestas en fase, es decir, el pico de onda de una se encuentra con el valle de la otra y así sucesivamente. El efecto es que las ondas se cancelan.
![007]()
Pueden darse casos intermedios en los que hay un descenso de la intensidad (amplitud) de la onda resultante.
Poniendo todo lo anterior junto
Imagina que tenemos dos electrones ahí en reposo y que hacemos pasar un haz de rayos X por ahí. Según hemos aprendido esos electrones describirán un movimiento oscilatorio y tendremos que emitirán ondas en casi todas las direcciones.
Pero tenemos dos electrones, es decir, ese proceso se repite dos veces por lo que tendremos ondas procedentes de un electrón y ondas procedentes del otro electrón. Esas ondas se superpondrán dando lugar a interferencias.
Suponiendo que el haz de rayos X inicial va de izquierda a derecha en este caso podemos ver qué pasa en diferentes direcciones respecto de la situación de los electrones. Podemos ver qué pasa en la misma dirección que la del haz inicial:
![008]()
Vemos que el pico de una onda coincide con el pico de la otra así que tendemos una interferencia constructiva. Si ponemos una pantalla sensible a los rayos X lo que veremos es que en sea dirección aparece una mancha.
Si subimos (o bajamos el ángulo) llegaremos a una dirección en la que tendremos esta situación:
![009]()
Ahí vemos claramente que esas dos ondas interfieren destructivamente. En la pantalla no aparecerá señal alguna.
Si seguimos subiendo (bajando) el ángulo volveremos a encontrar una interferencia constructiva:
![010]()
En la pantalla veremos otra señal de que hay llegado onda desde esa dirección. Lo que veríamos sería algo así:
![011]()
Una secuencia, un patrón, de regiones brillantes y regiones oscuras que son los puntos en los que hay direcciones donde ha habido interferencia constructiva y destructiva respectivamente.
¿Qué pasa si ponemos una fila de (muchos) electrones?
Pues pasará exactamente lo mismo pero muchas veces. Eso quiere decir que tendremos interferencias constructivas y destructivas. Pero cuantos más electrones tengamos mayor será la distancia entre puntos brillantes en la pantalla. El motivo se entiende con las figuras siguientes en las que representamos solo 3 electrones de todos los que tenemos en la fila.
![012]()
En la dirección en la que llega el haz tenemos interferencia constructiva, un punto brillante en la pantalla.
Pero si aumentamos el ángulo llegaremos a esta situación:
![013]()
Ahí vemos como la onda del primer electrón se cancela con la del segundo, la del tercer electrón se cancelará con la del cuarto, etc. Por tanto para volver a tener una interferencia constructiva y señal en la pantalla hemos de llegar a ángulos más grandes. Por tanto la separación entre puntos brillantes en la pantalla aumentará. Esto se ve en la siguiente figura con 2, 3, 4, 5 y 7 centros dispersores:
![014]()
Un detalle sobre estructuras
Cuando lanzamos rayos X sobre una estructura estos serán dispersados por los electrones de los átomos de la misma y se producirán los fenómenos que hemos explicado pero de diversas maneras. Lo que obtendremos será algo que se conoce como patrón de difracción.
![015]()
Veremos en la pantalla regiones brillantes (interferencias constructivas) y regiones oscuras (interferencias destructivas). El patrón será más o menos complicado y un tanto abstracto. Lo que no obtenemos es una imagen de la estructura interior del material por el que los rayos X han pasado. Para obtener una imagen deberíamos de disponer de una lente que recolectara toda la luz dispersada y la focalizara en la pantalla. Desgraciadamente aún no tenemos lentes que hagan eso de manera eficiente para los rayos X. Así que todo lo que tendremos será un patrón de difracción.
Supongamos que tenemos un cristal de sal que es una combinación ordenada de átomos (mejor iones) de sodio y cloro.
![salt1]()
En su interior los átomos de sodio y cloro se organizan en una red tridimensional:
![217734]()
Pero si hacemos pasar rayos X por el grano de sal lo que veremos será:
![nacl]()
De ahí se deduce la estructura del cristal indicada más arriba. ¿Cómo se hace eso? Bueno, la cuestión es que aunque nos parezca algo muy loco el patrón de difracción mostrado en la pantalla se relaciona matemáticamente con la estructura del material que lo genera. De hecho, eso que vemos ahí es la transformada de Fourier en el espacio de momentos de la estructura espacial del cristal (este comentario es para picar la curiosidad, nada más).
Lo que nos interesa aquí es un detalle curioso. Pensemos que tenemos un material con esta estructura:
![estruc1]()
El patrón de difracción será algo así:
![016]()
Ahora imaginemos que tenemos la habilidad de estirar las distancias entre los átomos de la estructura, todas ellas. ¿Cambiará el patrón de difracción?
La respuesta es sí y de una forma característica:
![estruc2]()
Es decir, hemos estirado las direcciones vertical y horizontal y el patrón de difracción cambia a lo siguiente:
![017]()
Las distancias en el patrón de difracción se han acortado en la dirección vertical y la dirección horizontal. Esta es una propiedad genérica de los patrones de difracción ya que hay una relación recíproca de distancias entre la red real y el patrón de difracción:
![018]()
Esto es conveniente tenerlo en mente.
Ahora sí, el aspecto de la foto 51
![001]()
Vamos a proceder a describir la foto 51.
Esta foto se consiguió haciendo pasar rayos X por una fibra compuesta por moléculas de ADN alineadas en un mismo eje:
![022]()
El patrón de difracción no es una única molécula de ADN es un promediado de la estructura completa de la muestra. Es decir, esta técnica no resuelve moléculas individuales sino que nos da una imagen “promedio” de lo que estemos iluminando. Captura la esencia de las moléculas presentes en la muestra. Esa foto es un recorte de un patrón que se repite en toda la pantalla del detector (que puede ser una placa fotográfica sensible a los rayos X).
Lo que vemos en la foto 51 son varias características más que evidentes.
1.- En el patrón hay una X formada por regiones oscuras discontinuas:
![019]()
2.- Las líneas discontinuas están en distancias fijas y tenemos 10 de estas líneas empezando en la línea central que se etiqueta como línea 0.
![020]()
Vemos que hay una línea que no está, la línea correspondiente al cuarto puesto (empezando por 0 en el centro de la X). Eso será muy importante.
En una foto más actual se ven mejor las distintas líneas:
![estruct]()
Y de ahí se obtiene la estructura del ADN.
![021]()
No te preocupes, no hemos acabado aún aunque estamos cerca.
Un par de detalles más
¿Qué pasa si nuestra muestra tiene una disposición de planos inclinados donde se sitúan los átomos que tienen los electrones que generan el patrón de difracción?
Lo que ocurre es lo siguiente:
![023]()
La orientación de la muestra real (línea oscura con bolas representando átomos) es recíproca al patrón de difracción resultante (manchas grises).
Si complicamos la situación y combinamos esas muestras reales en un zig-zag obtendremos algo así:
![024]()
Oh, sí, ahí hay una bonita X:
![025]()
Y lo que es más, podemos encontrar una relación entre los ángulos de las ramas del zig-zag y el ángulo en el patrón de difracción. Para ello hay que conocer la distancia de la muestra a la pantalla y la longitud de onda de los rayos X empleados. Aunque hace falta alguna información más, no es difícil encontrar la relación.
¿Qué tiene que ver esto con una hélice?
Bueno, la respuesta es simple, una hélice se puede ver así:
![026]()
O desde otro punto de vista:
![027]()
Esto se parece un poco más al zig-zag que hemos usado antes. Parece que el patrón de difracción de una hélice tiene toda la pinta de generar una X de algún modo.
Del patrón de difracción podemos obtener valiosa información de una hélice. Podemos encontrar la distancia entre giros de hélice, la P, el radio R y el ángulo de los brazos de la hélice.
Si tenemos una hélice continua el patrón de difracción será tal que así:
![028]()
Las manchas de las distintas líneas están separadas entre sí y eso está relacionado con la distancia de giro de hélice, con la P. Como sabemos la relación entre distancias es recíproca, así que si aumentamos la distancia de giro de hélice, aumentamos la P, esas manchas aparecerán más juntas:
![029]()
Si mantenemos la distancia de giro de hélice, P constante, pero aumentamos el radio de la hélice, el patrón será:
![030]()
El ángulo se cierra mucho. Por eso, midiendo el ángulo del patrón de difracción y conocida la distancia de la muestra a la pantalla y la longitud de onda es posible determinar el radio y ángulo de la hélice. La distancia de giro de hélice se determina midiendo la distancia entre las manchas.
Observar que en este caso las manchas del patrón de difracción son segmentos continuos. Eso es importante.
Por fin…
Volvamos a la foto 51 o a su versión más moderna:
![001]()
![estruct]()
Lo que podemos decir es que lo que está generando eso, el ADN, es una hélice.
Por otro lado, vemos que el patrón no tiene segmentos en los “escalones” sino que son más como manchas más localizadas. Eso es porque la hélice a la que nos enfrentamos no es continua sino que está compuesta por elementos que se repiten, los monómeros del polímero ADN, los nucleótidos.
Sabemos por lo tanto que en un giro de hélice tenemos 10 unidades de estos nucleótidos.
Conocidas las características del experimento, longitud de onda de los rayos X empleados, distancia de la muestra a la pantalla, etc, podemos determinar la distancia de paso de hélice, la P sin más que medir la distancia entre dos niveles consecutivos de manchas en el patrón de difracción. Si hiciéramos eso nos saldría que P=3.4nm.
¿Cómo de separados están los nucleótidos en el ADN? Pues si en la distancia P hay 10 de estas unidades estas estará separadas 0.34nm. Y además eso se puede medir midiendo la distancia del centro a la última capa del patrón de difracción. Recordad que las distancias pequeñas en la estructura real se transforman en grandes en el patrón de difracción y viceversa.
Al medir el ángulo de la X del patrón de difracción podemos determinar tanto el ángulo de los brazos de hélice y el radio de la misma.
Es gracias a esta foto que podemos determinar la estructura física del ADN.
![adnbien]()
¿Y la doble hélice?
Cierto, cierto.. aquí solo hemos hablado de una hélice simple. ¿Cómo se sabe que en realidad es una doble hélice?
Por esto:
![020]() Ahí faltan unas manchas que deberían de estar, las de la 4º capa. Y eso es lo que indica que es una doble hélice. La cuestión es que si hay dos hélices habrá posiciones en las que los átomos que generan la dispersión de los rayos X den lugar a una interferencia destructiva y por lo tanto faltarán señales que estarían en caso de ser una hélice simple.
Ahí faltan unas manchas que deberían de estar, las de la 4º capa. Y eso es lo que indica que es una doble hélice. La cuestión es que si hay dos hélices habrá posiciones en las que los átomos que generan la dispersión de los rayos X den lugar a una interferencia destructiva y por lo tanto faltarán señales que estarían en caso de ser una hélice simple.
No solo eso, si observamos la línea de la capa 6 veremos que es menos intensa de lo que se esperaría, eso es debido al mismo motivo. Por esos detalles sabemos que el ADN es una doble hélice.
Sin duda, este es uno de los grandes episodios de la historia de la ciencia y aunque conocer todos los detalles técnicos es ciertamente complejo podemos tener una ligera idea del secreto que esconde la foto 51.
Espero que os haya gustado… ¿Has votado a los chanchitos?
Nos seguimos leyendo…

. Por tanto la probabilidad de que salgan dos cincos a la vez es 1/36, la misma que de que salga primero un cuatro y luego un uno, porque sólo hay un resultado favorable.
, es igual a la multiplicación de sus probabilidades:
.
. En el caso particular en que todas las probabilidades son iguales, se tiene:
representa la velocidad de reacción. Para digerir este resultado podemos pasar a un ejemplo con números que nos clarifique la situación.
significa que el promedio de moléculas que reaccionarán en un segundo es 300, así que en un intervalo de tiempo Δt = 0,01 es esperable que reaccionen
moléculas.
moléculas.
es el número de moléculas que esperamos que reaccionen, pero desde luego en todos los intervalos de tiempo no será así. A veces serán algunas más y a veces algunas menos. El error en los modelos deterministas es dar por hecho que en todo intervalo reaccionan
moléculas.
(indicando
la reacción correspondiente) después de que transcurra un tiempo
sin que haya habido ningún tipo de reacción.
sin reacciones.
a la suma de las velocidades de todas las reacciones:
. Para hacer esto dividimos el intervalo
en muchos intervalos pequeños de tiempo de duración
(con lo que tendremos en total
intervalos).
.
sin reacciones es la multiplicación de ambas probabilidades:
es
y
?. ¿Cómo se trabaja con ellas? La respuesta en la tercera y última parte de la entrega.







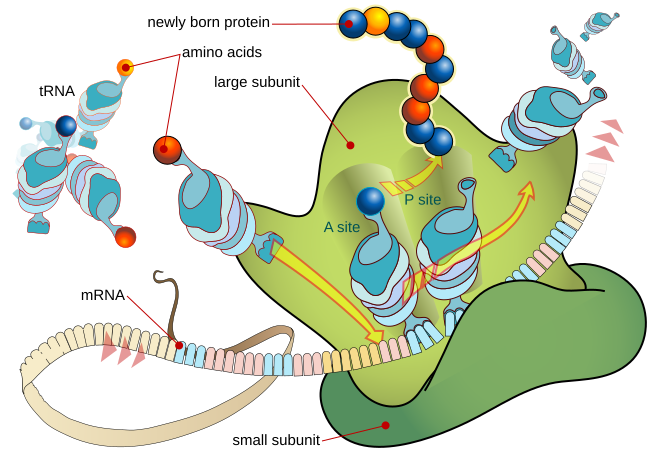
 En este blog hemos hablado en alguna ocasión de la importancia de las
En este blog hemos hablado en alguna ocasión de la importancia de las 


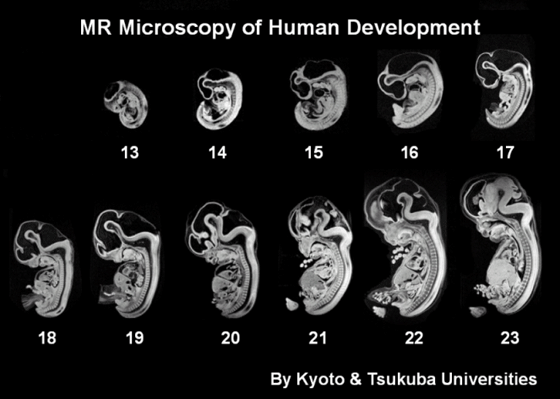
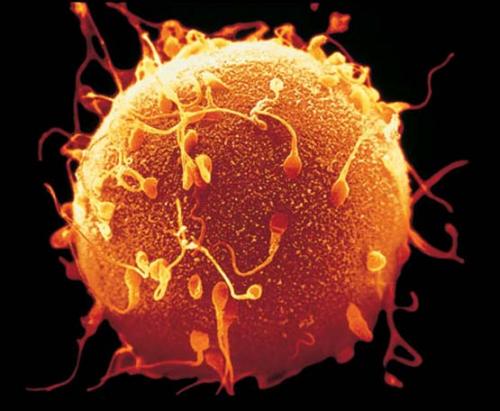 ¿Cómo a partir de un óvulo fecundado sale un ser humano? ¿Cómo saben las células, que no son más que subdivisiones del óvulo, cómo se tienen que diferenciar para dar lugar a diferentes tejidos en diferentes posiciones? Este tema siempre me ha vuelto loco, me parece que es uno de los problemas más fascinantes de la ciencia.
¿Cómo a partir de un óvulo fecundado sale un ser humano? ¿Cómo saben las células, que no son más que subdivisiones del óvulo, cómo se tienen que diferenciar para dar lugar a diferentes tejidos en diferentes posiciones? Este tema siempre me ha vuelto loco, me parece que es uno de los problemas más fascinantes de la ciencia.























































































![An American Tail [1986] [DVD5-R1] [Latino]](http://iili.io/FjktrS2.jpg)

